Comment séparer des cercles imbriqués les uns dans les autres ?
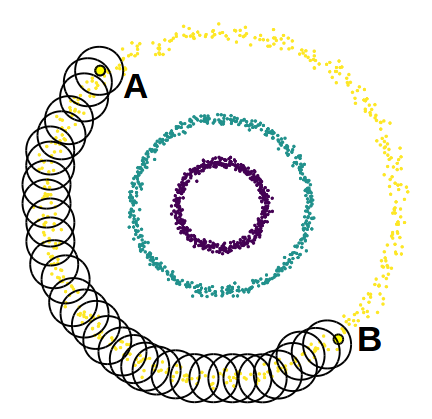
Quel algorithme de clustering utiliser pour former des clusters non convexes, comme des demi-lunes ou des cercles imbriqués les uns dans les autres ? Dans la partie précédente, nous avons vu que le k-means forme nécessairement des clusters convexes, ce qui n'est pas le cas de ce genre de figure. Par exemple, sur l'image ci-dessous, un segment reliant un point en haut du cercle extérieur à un point en bas du même cercle pourra contenir des points des deux autres cercles/clusters.

Nous avons vu précédemment que le kernel k-means permet de résoudre ce genre de problème. Mais ce n'est pas la seule façon de faire !
Clustering par densité
Les méthodes de clustering par densité sont basées sur l'observation suivante : bien que les deux points que j'ai mentionnés plus haut (notés A et B sur la figure ci-dessous) ne puissent pas être liés l'un à l'autre par un segment sans intersecter les deux autres clusters, on peut créer un chemin pour passer de l'un à l'autre de proche en proche en restant à l'intérieur du même cluster.
Un peu de vocabulaire maintenant...
Étant donné un nombre réel positif , on appelle epsilon-voisinage d'un point x l'ensemble des points du jeu de données dont la distance à x est inférieure à :
Étant donné un entier naturel , on dit que x est un point intérieur (core point, en anglais) si son -voisinage contient au moins points : .
On dit maintenant que deux points u et x sont connectés par densité si l'on peut passer de l'un à l'autre par une suite d' -voisinages contenant chacun au moins points. Autrement dit, il existe une suite de points intérieurs tels que appartient à l' -voisinage de u, appartient à l' -voisinage de , et ainsi de suite, jusqu'à ce que x appartienne au voisinage de . On dit aussi alors que x est atteignable par densité depuis u.
Pourquoi tout ce vocabulaire ? Parce que nous allons maintenant créer des clusters de points atteignables par densité les uns depuis les autres.
DBSCAN
L'algorithme DBSCAN (Density-Based Spatial Clustering of Applications with Noise) a été introduit en 1996 dans ce but. Cet algorithme est très utilisé, raison pour laquelle il a obtenu en 2014 une distinction de contribution scientifique ayant résisté à l'épreuve du temps.
DBSCAN itère sur les points du jeu de données. Pour chacun des points qu'il analyse, il construit l'ensemble des points atteignables par densité depuis ce point : il calcule l'epsilon-voisinage de ce point, puis, si ce voisinage contient plus de n_min points, les epsilon-voisinages de chacun d'entre eux, et ainsi de suite, jusqu'à ne plus pouvoir agrandir le cluster. Si le point considéré n'est pas un point intérieur, c'est-à-dire qu'il n'a pas suffisamment de voisins, il sera alors étiqueté comme du bruit. Cela permet à DBSCAN d'être robuste aux données aberrantes, puisque ce mécanisme les isole.
L'algorithme de DBSCAN est le suivant :
1. Prendre un point x qui n’a pas été visité
2. Construire N = voisinage(eps, x)
3. If |N| < n_min:
Marquer x comme bruit
Else:
Initaliser C = {x}
agrandir_cluster(C, N, eps, n_min)
Ajouter C à la liste des clusters
Marquer tous les points de C comme visités
4. Repeat 1-3 until tous les points ont été visitésLa procédure agrandir_cluster est donnée par :
agrandir_cluster(C, N, eps, n_min):
For u in N:
If u n’a pas été visité:
N’ = N(eps, u)
If |N’| >= n_min:
N = union(N, N’)
If u n’appartient à aucun autre cluster:
Ajouter u à CAvantages et inconvénients de DBSCAN
L'algorithme DBSCAN est difficile à utiliser en très grande dimension : souvenez-vous du fléau de la dimensionalité. Les boules de rayon epsilon et de grande dimension ont tendance à ne contenir aucun autre point.
Le choix des paramètres et peut aussi être délicat : il faut veiller à utiliser des paramètres qui permettent de créer suffisamment de points intérieurs (ce qui n'arrivera pas si est trop grand ou trop petit). En particulier, cela signifie que DBSCAN ne pourra pas trouver des clusters de densités différentes.
DBSCAN a le grand avantage d'être efficace en temps de calcul sans requérir de prédéfinir le nombre de clusters. Enfin, comme je l'ai dit au début de ce chapitre, il permet de trouver des clusters de forme arbitraire.
